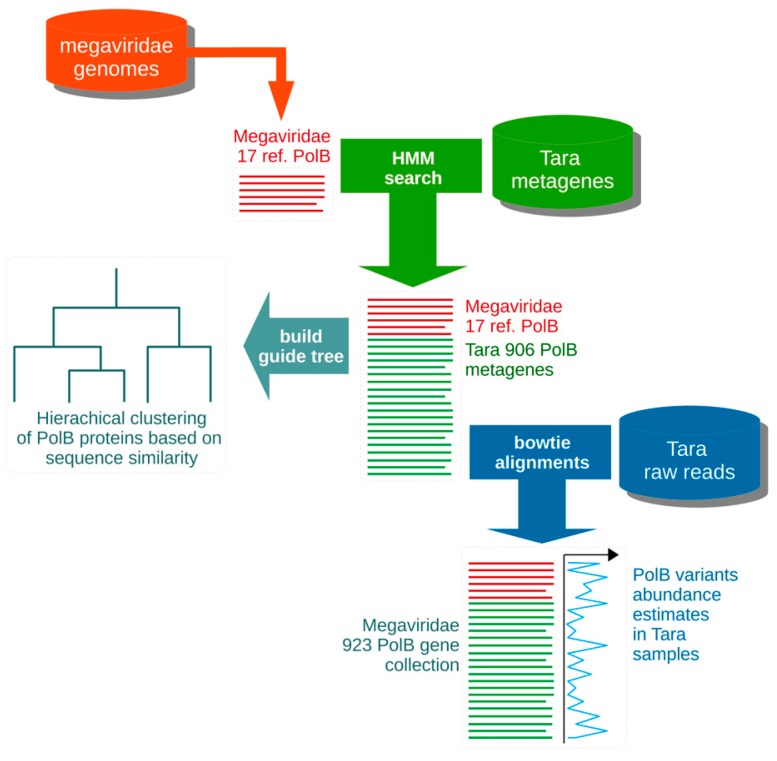
La diversité des virus géants à ADN double brin est très élevée, mais ils sont particulièrement difficiles à observer et à cultiver. En collaboration avec mes collègues de l’université de Kyoto, j’ai développé un workflow bioinformatique pour la conception d’un jeu d’amorces ciblant l’ensemble des membres connus des mégaviridae. Le workflow intitulé UjiLity, en rapport avec le campus d’Uji à Kyoto où j’ai eu le privilège de le développer, identifie la sous région la plus conservée du jeu de données cible en respectant les contraintes spécifiées par l’utilisateur, notamment la longueur de l’amplicon et des oligonucléotides, leur température de fusion très précise, ainsi que la dégénérescence maximale souhaitée. Le défi spécifique a été de surmonter l’énorme diversité des séquences cibles, en permettant de définir plusieurs paires d’amorces.
Le jeu d’amorces que j’ai conçu à cette occasion avec le workflow UjiLity a été testé sur des échantillons d’eau de mer, et à démontré que cette approche permet d’estimer avec beaucoup de profondeur la diversité des virus géants dans des échantillons environnementaux. Pour en savoir plus, se référer à Viruses 2018 « Degenerate PCR Primers to Reveal the Diversity of Giant Viruses in Coastal Waters ». Le code source et les données sont disponibles sur https://github.com/PHingamp/UjiLity .